
《肿瘤》

【飞桨开发者说】韩霖,PPDE飞桨开发者技术专家,吉林大学计算机科学与技术学院,主要研究医学影像方向。

项目背景
近年来,快速发展的深度学习技术已经渗透进了各行各业,医疗方面也不例外。这篇文章我主要介绍如何使用深度学习计算机视觉方法对CT扫描中的肝脏和肝脏肿瘤进行分割。
根据2018年的统计数据[1],肝脏肿瘤是全球第7常见的肿瘤,但致死病例总数却在所有肿瘤类疾病中排名第二。早发现早治疗能有效提升肝脏肿瘤疾病的治愈率,但人工在大量的肝脏CT影像中寻找体积很小的肿瘤工作量极大,也很容易漏检。这个场景下,使用深度学习算法自动进行快速、准确的肝脏及肝脏肿瘤分割筛查是一个很好的解决方案。
基于飞桨PaddlePaddle框架,我使用Res-Unet网络结构在 LiTS 数据集[2]上训练了一个分割网络,最终在肝脏和肝肿瘤上分别达到了 0.92 和 0.77 的分割准确率。LiTS数据集是目前最大的开源肝脏分割数据集,其中包含130名患者的CT扫描和医生对患者肝脏及肿瘤的分割标注,下图是数据集中的一个示例:
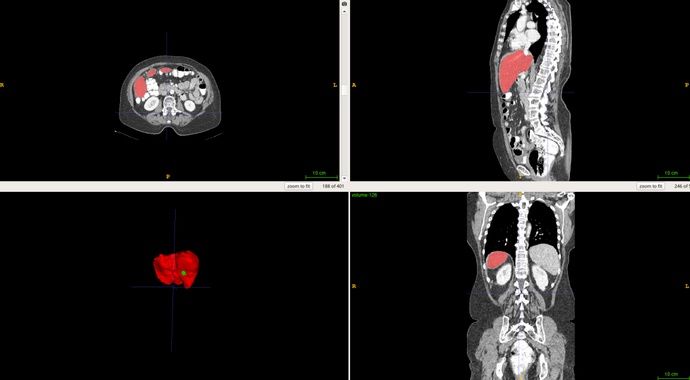
图1 肝脏分割示例
项目在AI Studio上公开,提供包含数据集在内的完整环境,fork后可以直接运行。
此外还有更适合命令行执行的Github开源项目medSeg,经过性能优化,训练及推理速度更快。
网络结构介绍
本文中主要针对项目使用的网络结构,数据预处理及增强,Loss,训练和推理步骤进行描述。
首先简单介绍项目中用到的网络结构Res-Unet。在医学影像领域,Unet[3]结构因为其网络参数规模较小,实现简单,边界分割比较准确被广泛应用。其结构如下图所示:
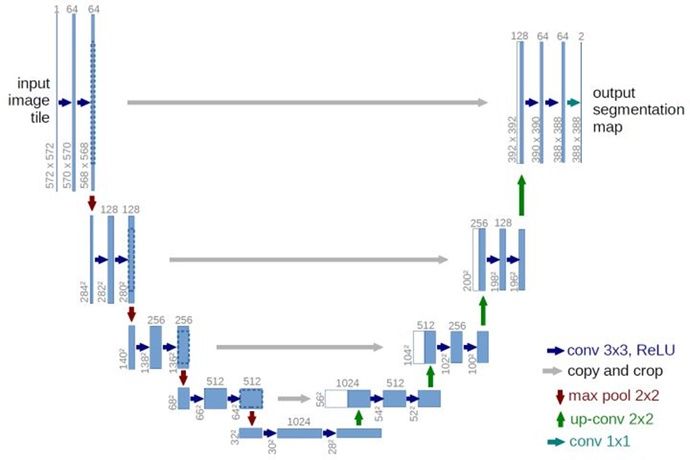
图2 Unet网络结构
其采用编码器-解码器结构,是一个 U 的形状,因此作者取名Unet。网络首先对输入图片进行了左边的4组卷积和下采样操作来获取图像的抽象特征,之后通过右边的对称的4组反卷积和上采样将图像放大回接近输入图像的大小。Unet的一个重要创新是在相同深度的下采样和上采样操作之间加入了跳转连接(图中横向灰色箭头所示),有效地提升了网络的分割精度。具体的实现方法一般是将左侧卷积block的输出拼接到右侧同一深度反卷积block的输入上。
这样反卷积block的输入特征图大小不变,但是厚度变成了原来的两倍。其中一半是绿色箭头代表的下层反卷积block的输入,给网络提供更抽象的高阶图像特征;另一半是灰色箭头代表的左侧卷积block的输出,给网络提供更准确的位置信息,提升边缘分割精度。
我使用的Res-Unet网络在Unet结构的基础上引入了残差连接,如下图所示。具体的做法是添加一条从两次卷积的输入到输出的连接,并做一次卷积操作。这种残差结构改善了网络的梯度流通,避免网络退化,并能加速网络收敛。
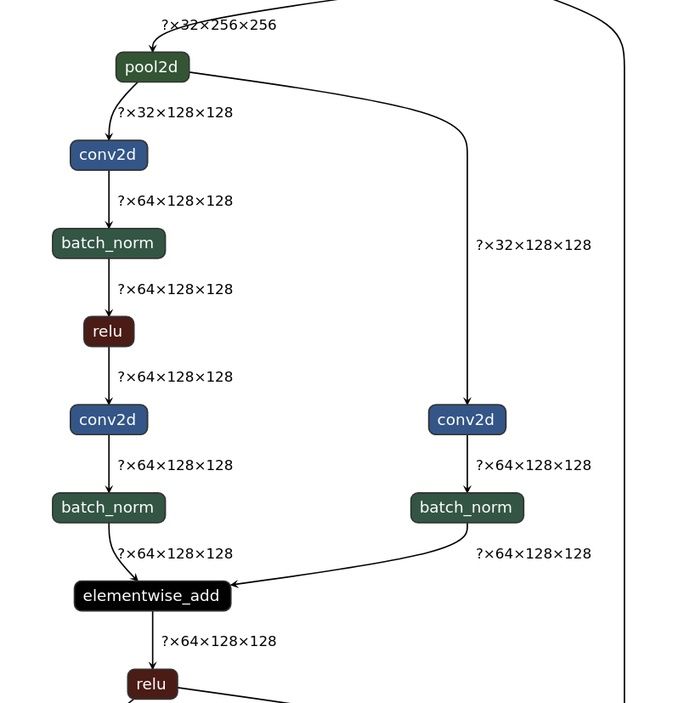
图3 残差连接
具体的网络构建代码比较复杂,这里不做详细展示,可以访问AI Studio项目或Github repo查看。
数据处理及增强
上述的Res-Unet结构是一个2D的分割网络,因此我们首先将LiTS数据集中3D的CT扫描分成2D的切片。CT在拍摄和重建的过程中会引入一些噪声,因此我们只保留-1024到1024范围内的数据。经过这两步处理,可以得到大概1万张CT扫描切片及对应的分割标签,随机选择一组进行可视化结果如下

图4 2D切片
在训练深度神经网络的过程中,我们通常需要在训练集上训练多个epoch以让网络达到一个比较高的训练准确率。但是这样做又容易使网络过拟合训练集,其表现为网络在训练集上准确率很高但是测试时准确率偏低。针对这个问题有多方面的解决方案,数据增强是其中重要的一种。这个项目中我们采用的数据增强策略包括随机水平、垂直翻转、随机旋转、随机尺度缩放、随机位置裁剪和弹性形变。在项目中可以看到具体代码,图5是对图4中数据进行数据增强的结果:
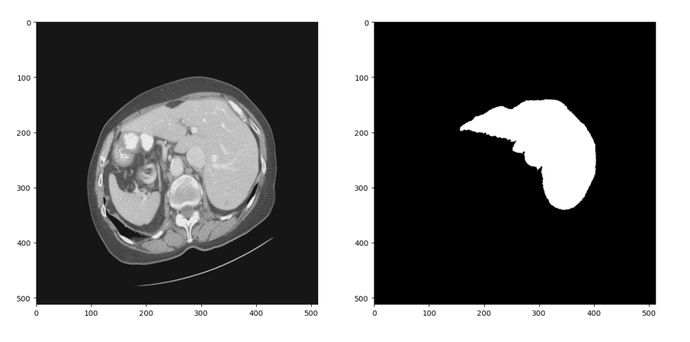
图5 数据增强效果
CT图像和分割标签共同进行了左右翻转,逆时针15度旋转,0.8倍尺度缩放和弹性形变。虽然一些简单的数据增强步骤过后图像看起来没有很大区别,但是只要图像有变化对算法来说就是新的数据,结合Droupout、权重正则化等方法能较好地抑制网络过拟合,提升测试准确率。
上一篇:当恶性肿瘤来临时,身体会暴露出‘’告密者’‘
下一篇:没有了